來源:nature Science
編輯:Priscilla 好困
【新智元導(dǎo)讀】昨日,DeepMind和華盛頓大學(xué)分別在nature和Science兩大頂級(jí)雜志發(fā)布了各自預(yù)測(cè)蛋白質(zhì)結(jié)構(gòu)的工具,并同時(shí)開源了代碼。
nature和Science兩本雜志一直相愛相殺,總是喜歡爭(zhēng)著發(fā)表科學(xué)領(lǐng)域中的重大發(fā)現(xiàn)、重要突破,搶奪大師文章的發(fā)表版權(quán)。
這次也不例外。
7月15日,DeepMind在Nature上發(fā)表了一篇論文,開源了其基于深度學(xué)習(xí)神經(jīng)網(wǎng)絡(luò)的AlphaFold 2模型。

論文鏈接:https://www.nature.com/articles/s41586-021-03819-2
開源代碼:https://github.com/deepmind/alphafold
與此同時(shí),在隔壁的Science上,華盛頓大學(xué)也發(fā)表了自己開發(fā)的蛋白質(zhì)預(yù)測(cè)工具RoseTTAFold。RoseTTAFold不僅性能上和AlphaFold 2相當(dāng),預(yù)測(cè)蛋白質(zhì)結(jié)構(gòu)也「快、狠、準(zhǔn)」。

論文鏈接:https://science.sciencemag.org/content/early/2021/07/14/science.abj8754
開源代碼:https://github.com/RosettaCommons/RoseTTAFold
當(dāng)然,RoseTTAFold最厲害的一點(diǎn)在于,其端到端版本在RTX2080上生成少于400個(gè)殘基的蛋白質(zhì)的骨干坐標(biāo)只需要10分鐘。
這顯然讓成千上萬的生物學(xué)家松了一口氣,畢竟一個(gè)2080還是能買得到的。
隨著RoseTTAFold和AlphaFold 2的代碼開源,有研究人員希望能在備受關(guān)注的兩個(gè)領(lǐng)域方面取得新的進(jìn)展:
- 預(yù)測(cè)多個(gè)相互作用的蛋白質(zhì)的復(fù)合體結(jié)構(gòu);
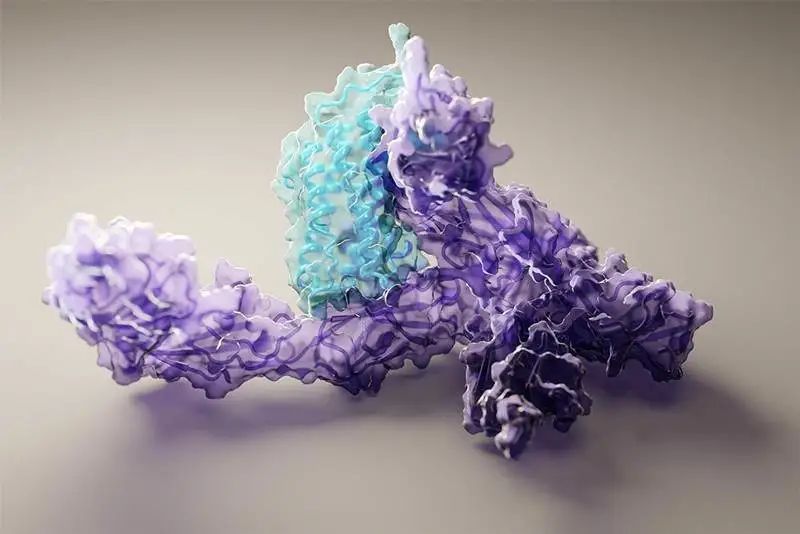
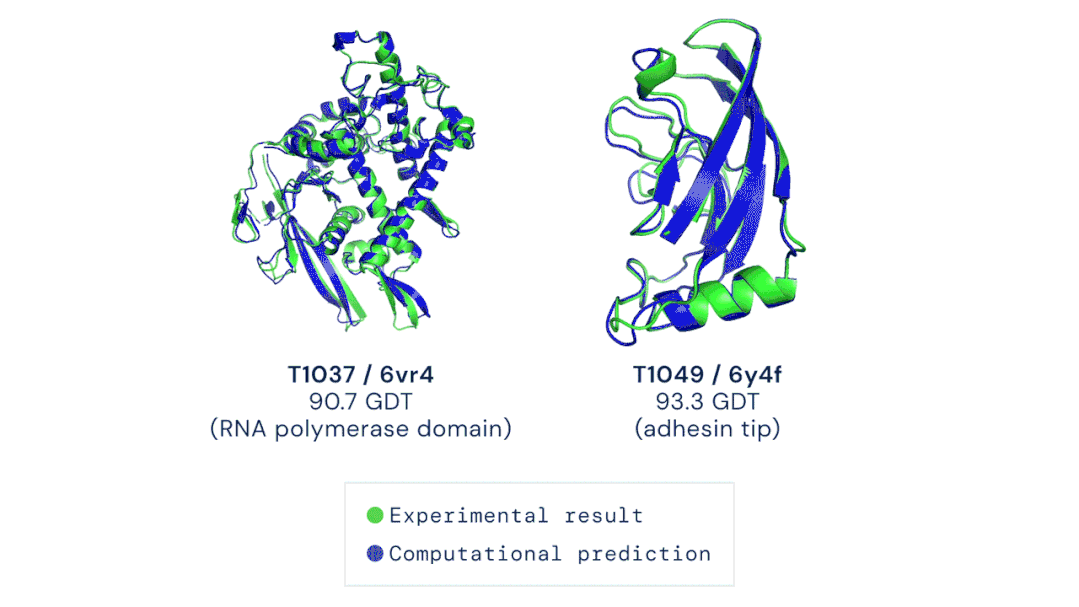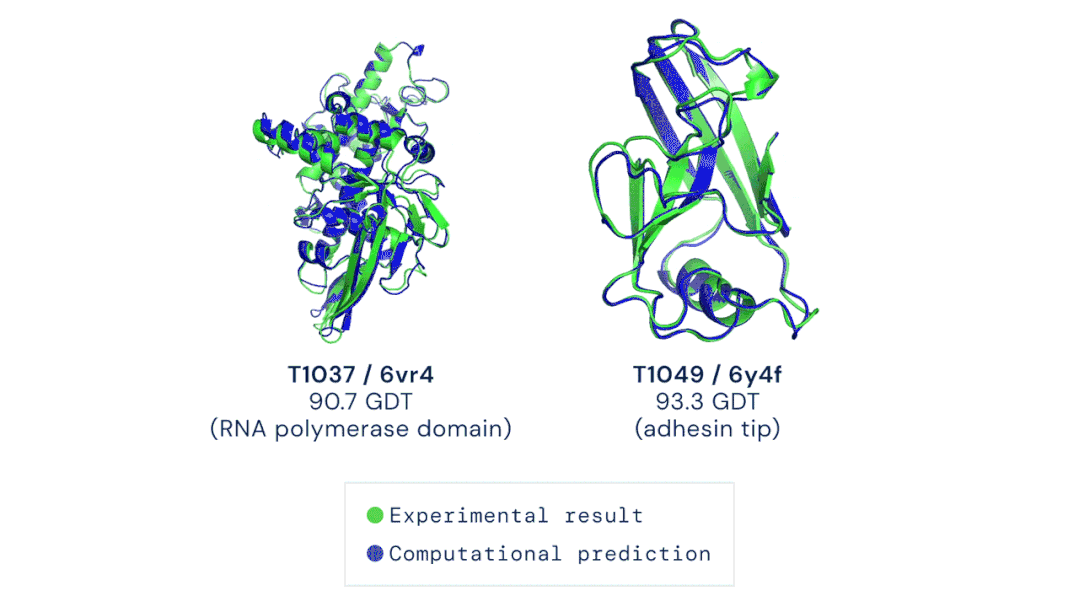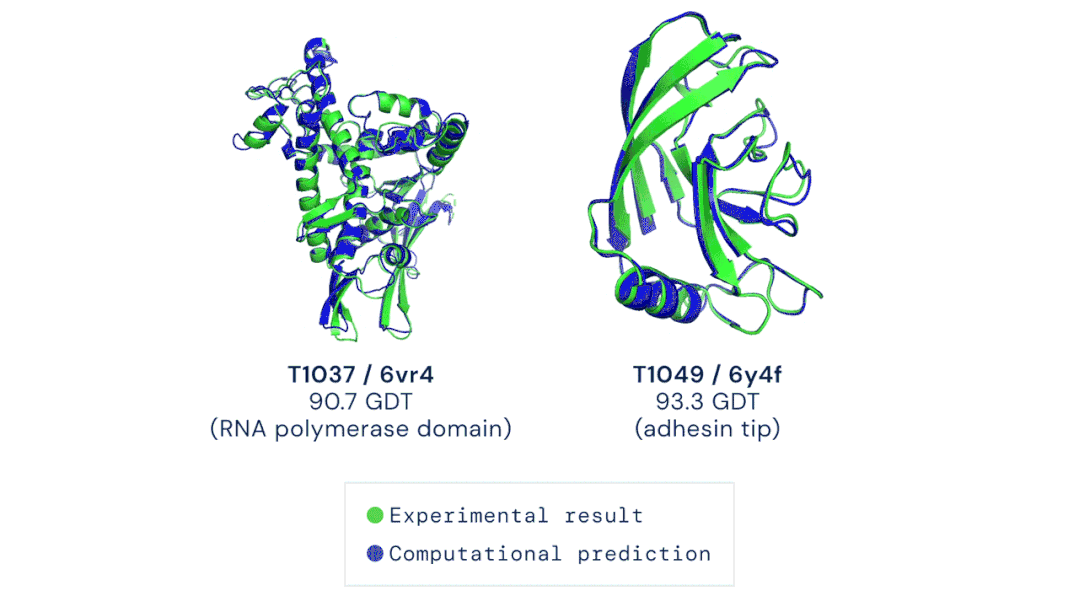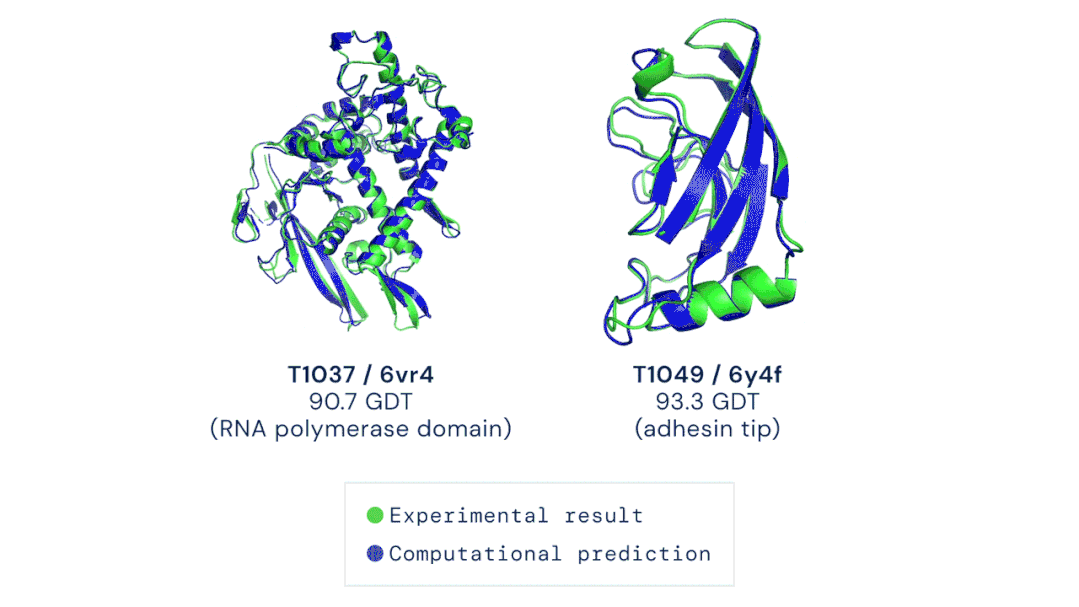
論文的其中一位作者表示,很高興自己能夠?yàn)檫@個(gè)巨大的里程碑做出貢獻(xiàn)。DeepMind的創(chuàng)始人兼CEO表示,很期待開源代碼之后能給更多研究帶來的可能性。Facebook AI的田淵棟則從AI的角度發(fā)表了對(duì)AlphaFold的看法:蛋白質(zhì)是生命的物質(zhì)基礎(chǔ),是構(gòu)成細(xì)胞的基本有機(jī)物,也是生命活動(dòng)的主要承擔(dān)者。蛋白質(zhì)由氨基酸通過「脫水縮合」的方式組成多肽鏈,經(jīng)過盤曲折疊,形成具有一定空間結(jié)構(gòu)的物質(zhì)。不同空間結(jié)構(gòu)也就決定了蛋白質(zhì)的不同功能。一直以來,「蛋白質(zhì)折疊問題」都是生物學(xué)中的一個(gè)巨大挑戰(zhàn)。如果用暴力計(jì)算法列舉出一個(gè)典型蛋白質(zhì)的所有可能構(gòu)型,所需的時(shí)間可能比已知的宇宙年齡還要長(zhǎng)。生物醫(yī)學(xué)領(lǐng)域?yàn)榱颂剿鞯鞍踪|(zhì)復(fù)雜的3D結(jié)構(gòu),只能通過低溫電子顯微鏡(CryoEM)、核磁共振或X射線晶體學(xué)等手段,經(jīng)過大量試錯(cuò)才能最終確定蛋白質(zhì)的結(jié)構(gòu)。理論上,一個(gè)蛋白質(zhì)在確定其最終結(jié)構(gòu)前,可能的折疊方式的數(shù)量是天文數(shù)字。1969年,Cyrus Levinthal指出,通過計(jì)算列舉一個(gè)典型蛋白質(zhì)的所有可能構(gòu)型,需要的時(shí)間比已知宇宙的年齡還要長(zhǎng)——可能有10^300種構(gòu)型。因此,這些方法有一個(gè)巨大的缺陷:耗費(fèi)大量的時(shí)間和精力。為解決這一困難,DeepMind利用人工智能,開發(fā)了一個(gè)能夠預(yù)測(cè)蛋白質(zhì)結(jié)構(gòu)的系統(tǒng):AlphaFold。2018年,AlphaFold在國際蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)競(jìng)賽(CASP)上首次亮相,獲得了馬克斯·普朗克發(fā)展生物學(xué)研究所(Max Planck Institute for Developmental Biology)所長(zhǎng)、CASP評(píng)估員Andrei Lupas的高度評(píng)價(jià):「AlphaFold驚人的精確模型幫助我們解決一直困擾科學(xué)界的蛋白質(zhì)結(jié)構(gòu)問題,重新啟動(dòng)了學(xué)界的研究。」預(yù)測(cè)的人類白細(xì)胞介素-12蛋白與其受體結(jié)合的結(jié)構(gòu)圖預(yù)測(cè)蛋白質(zhì)的3D結(jié)構(gòu)則有著相當(dāng)重要的作用。其能夠幫助科學(xué)家發(fā)現(xiàn)蛋白質(zhì)功能失調(diào),以及導(dǎo)致某些疾病的原因,這就為藥物的研發(fā)提供了一條全新的途徑,從而能夠快速地進(jìn)行醫(yī)療治療。DeepMind的AlphaFold 2模型在2020年的蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)比賽(CASP)取得了出色的成績(jī)。比賽的評(píng)分中位數(shù)達(dá)到了92.4 GDT。也就是預(yù)測(cè)的均方根誤差約為1.6埃,相當(dāng)于一個(gè)原子的寬度(或0.1納米)。即使是最難的蛋白質(zhì)目標(biāo),評(píng)分中位數(shù)也能達(dá)到87.0GDT。AlphaFold 2可以在不到10分鐘的時(shí)間內(nèi)預(yù)測(cè)出接近晶體結(jié)構(gòu)精度的蛋白質(zhì)結(jié)構(gòu)。對(duì)一個(gè)超過2千殘基的病毒RNA聚合酶的預(yù)測(cè)研究人員對(duì)AlphaFold2方法的進(jìn)展進(jìn)行了概括:從多個(gè)序列排列(MSA)開始,而不是從MSA得出的反協(xié)方差矩陣等更多處理過的特征開始;
用注意力機(jī)制取代二維卷積,更好地代表沿序列遠(yuǎn)處殘基之間的相互作用;
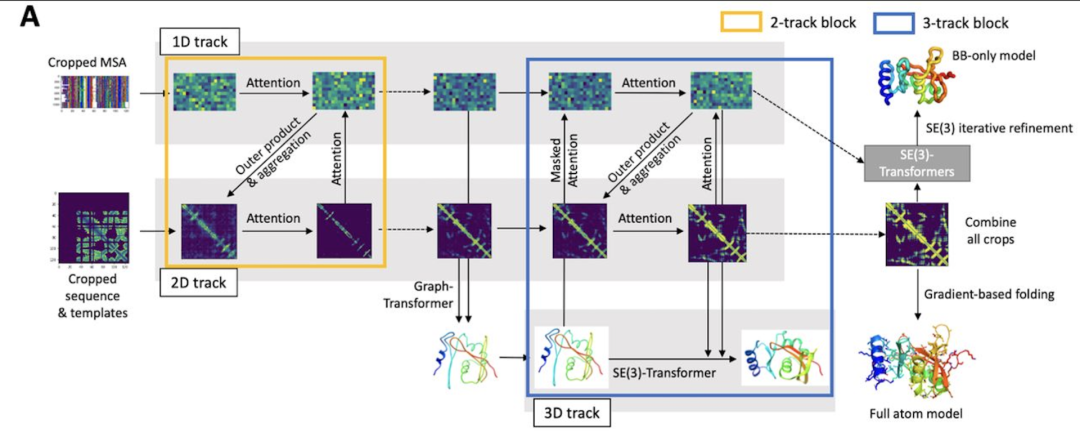
使用雙軌網(wǎng)絡(luò)結(jié)構(gòu),其中一維序列水平和二維距離圖水平的信息被迭代轉(zhuǎn)換并來回傳遞;
使用SE(3)-Transformer網(wǎng)絡(luò)來直接完善由雙軌網(wǎng)絡(luò)生成的原子坐標(biāo)(而不是像以前的方法那樣的二維距離圖);
端到端學(xué)習(xí),其中所有網(wǎng)絡(luò)參數(shù)通過反向傳播從最終生成的三維坐標(biāo)通過所有網(wǎng)絡(luò)層回到輸入序列來優(yōu)化。
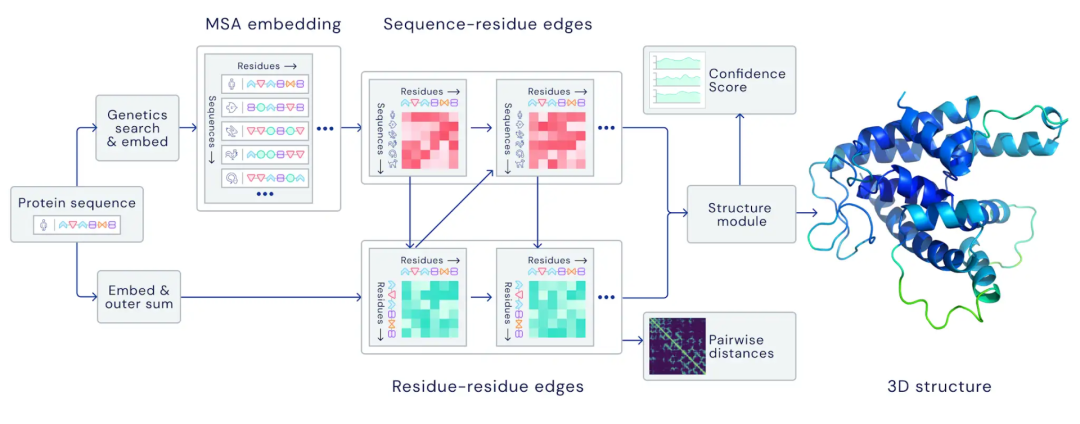
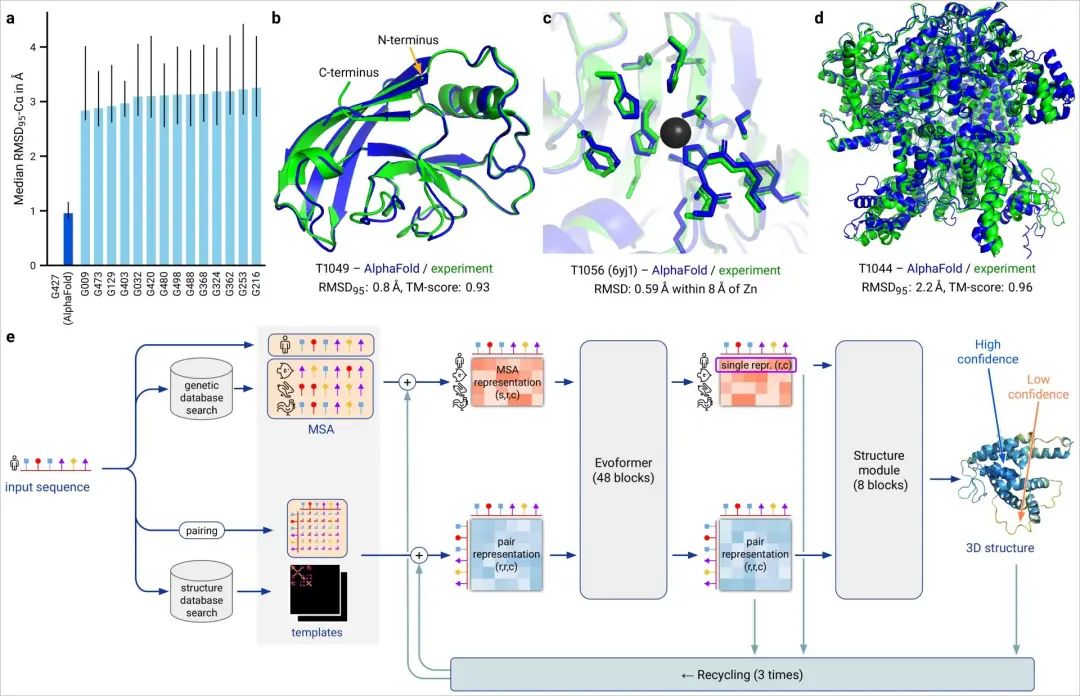
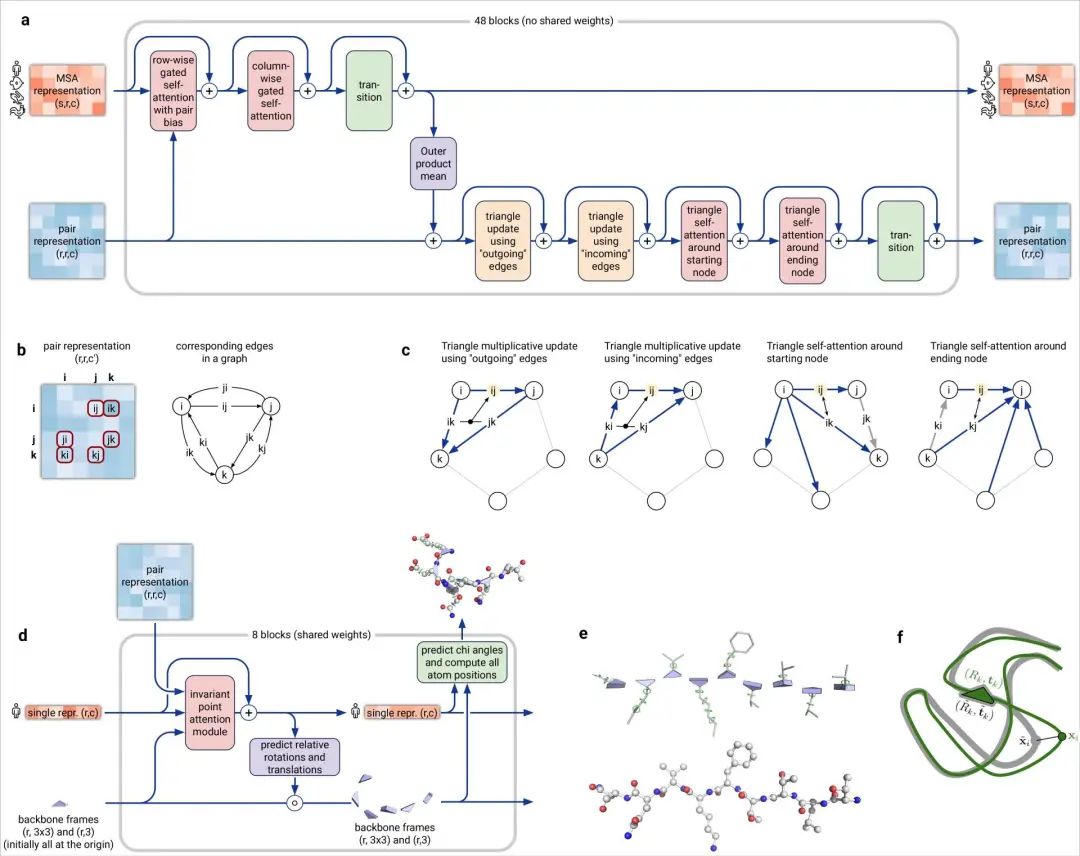
DeepMind表示,AlphaFold 2可以對(duì)蛋白質(zhì)的基本物理結(jié)構(gòu)進(jìn)行十分準(zhǔn)確的預(yù)測(cè),并能夠在幾天內(nèi)生成高精度的結(jié)構(gòu)。此外,模型還能利用內(nèi)部的內(nèi)部置信度來預(yù)測(cè)每個(gè)預(yù)測(cè)的蛋白質(zhì)結(jié)構(gòu)中哪些部分是可靠的。訓(xùn)練數(shù)據(jù)來自大約17萬個(gè)蛋白質(zhì)結(jié)構(gòu),以及包含未知結(jié)構(gòu)的蛋白質(zhì)序列的大型數(shù)據(jù)庫。期間DeepMind使用了16個(gè)TPU進(jìn)行訓(xùn)練(即128個(gè)TPUv3核心或大致相當(dāng)于約100-200個(gè)GPU)。神經(jīng)網(wǎng)絡(luò)模型結(jié)構(gòu)其中,模型對(duì)蛋白質(zhì)序列以及氨基酸殘基對(duì)進(jìn)行操作,在兩種表征之間迭代傳遞信息以生成結(jié)構(gòu)。AlphaFold生成的蛋白質(zhì)高精度結(jié)構(gòu)
RoseTTAFold:媲美AlphaFold 2的預(yù)測(cè)工具
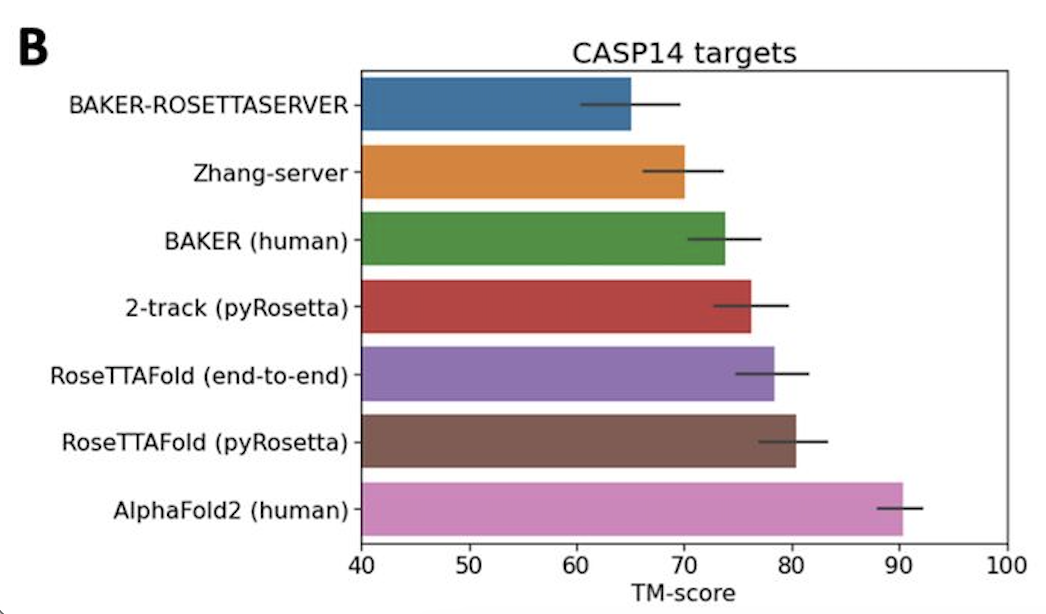
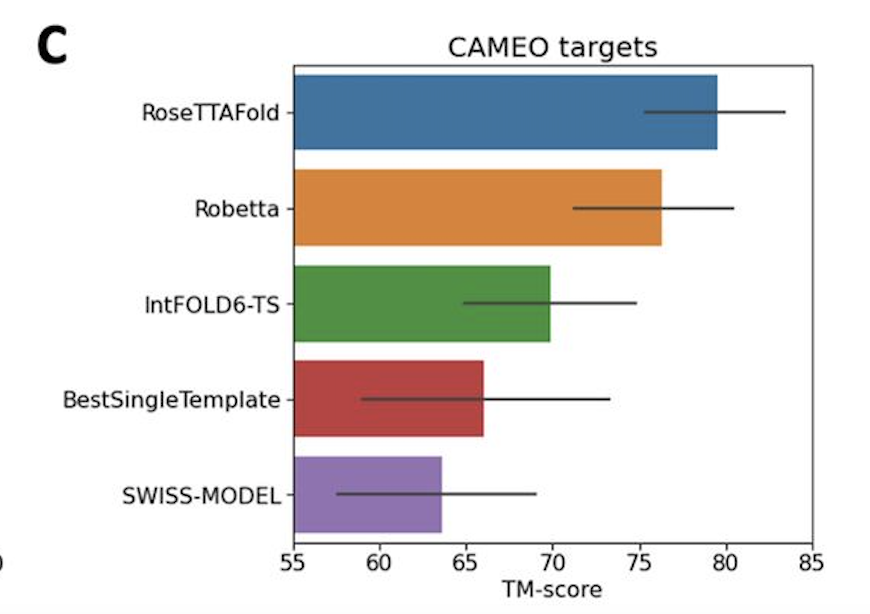
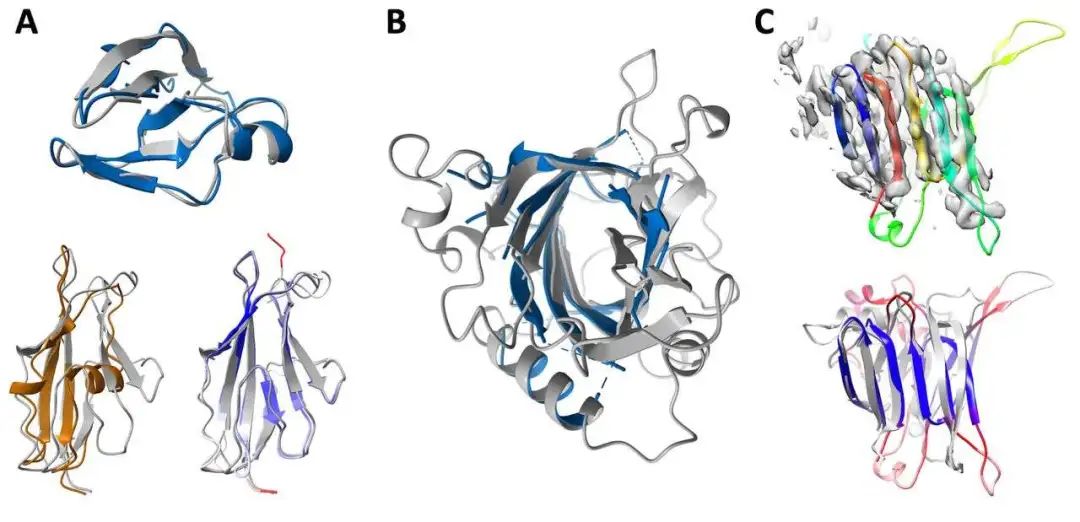
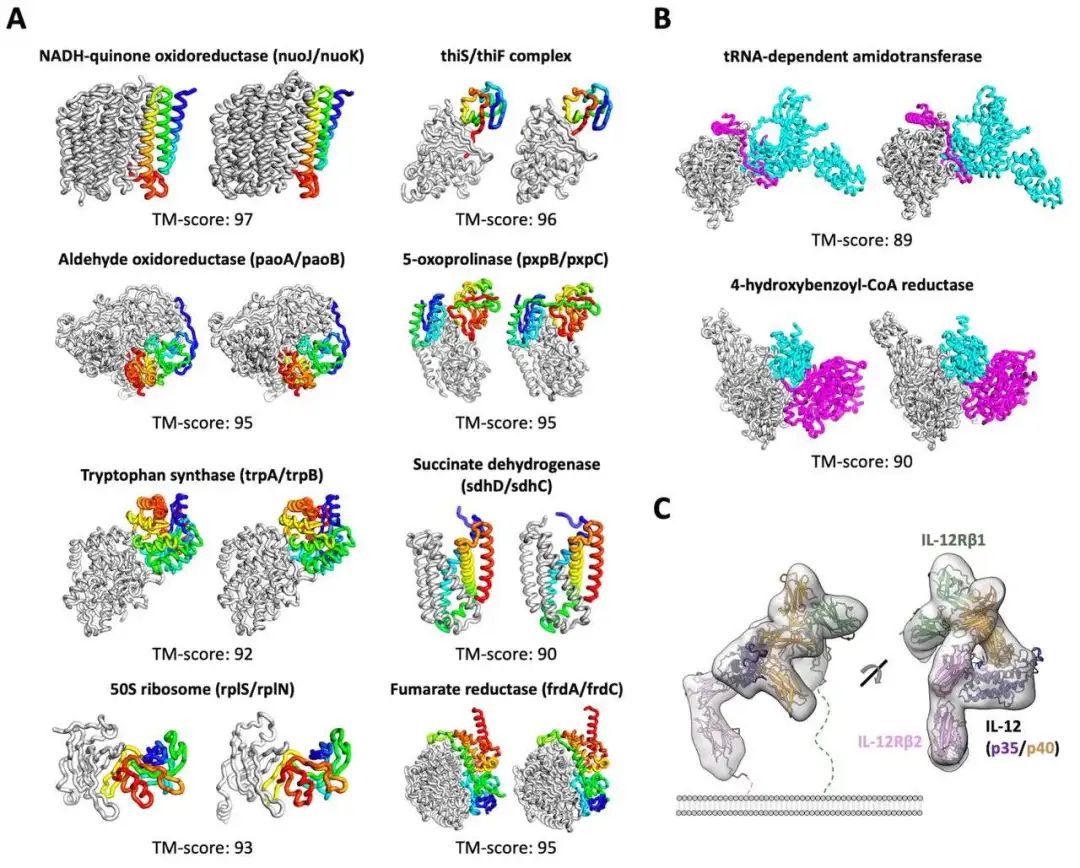
無獨(dú)有偶,隔壁Science雜志也刊登了另一個(gè)蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)的工具,名叫「RoseTTAFold」,特點(diǎn)是「快、準(zhǔn)、狠」。2020年,DeepMind在CASP 14大會(huì)上介紹了它在該蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)上的顯著進(jìn)展。華盛頓大學(xué)醫(yī)學(xué)院蛋白質(zhì)設(shè)計(jì)研究所的研究人員看在眼里,受其啟發(fā),與哈佛、劍橋、德克薩斯大學(xué)西南醫(yī)學(xué)中心、勞倫斯伯克利國家實(shí)驗(yàn)室聯(lián)手,共同研發(fā)了一款基于深度學(xué)習(xí)的蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)工RoseTTAFold。這款工具利用深度學(xué)習(xí),僅憑有限的信息,就能在普通游戲本上快速而準(zhǔn)確地預(yù)測(cè)蛋白質(zhì)結(jié)構(gòu),在短時(shí)間就能構(gòu)建出復(fù)雜的生物組建模型。目前,該研究團(tuán)隊(duì)已經(jīng)用RoseTTAFold計(jì)算出了數(shù)百種新的蛋白質(zhì)結(jié)構(gòu),其中就包括了許多鮮為人知的人類基因組蛋白。RoseTTAFold預(yù)測(cè)出了與脂質(zhì)代謝問題、炎癥紊亂和癌細(xì)胞生長(zhǎng)相關(guān)的蛋白質(zhì)結(jié)構(gòu)。RoseTTAFold是一個(gè)「三軌」神經(jīng)網(wǎng)絡(luò)("three-track" neural network),它能夠兼顧蛋白質(zhì)序列模式、氨基酸如何相互作用以及蛋白質(zhì)三維結(jié)構(gòu)的可能性。在這個(gè)架構(gòu)中,信息在一維(氨基酸序列)、二維(距離)和三維(坐標(biāo))之間來回流動(dòng),從而能夠集中推理出蛋白質(zhì)化學(xué)部分與折疊結(jié)構(gòu)之間的關(guān)系。RoseTTAFold 架構(gòu)包含一、二、三維注意力軌道,軌道之間信息能夠來回流動(dòng)在CASP14目標(biāo)取得的平均TM-score在CAMEO實(shí)驗(yàn)中取得的盲基準(zhǔn)結(jié)果蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)不斷取得新進(jìn)展,最關(guān)鍵的問題莫過于:能夠使用什么準(zhǔn)確的蛋白質(zhì)結(jié)構(gòu)模型?團(tuán)隊(duì)研究了RoseTTAFold通過X射線晶體學(xué)和低溫電子顯微鏡,研究了促進(jìn)實(shí)驗(yàn)結(jié)構(gòu)測(cè)定實(shí)用性,為目前為之結(jié)構(gòu)的關(guān)鍵蛋白質(zhì)提供模型。RoseTTAFold方法的準(zhǔn)確性遠(yuǎn)高于現(xiàn)有方法,因此,研究人員希望能夠測(cè)試出這款工具是否能夠解決分子置換(MR)這一從未解決的問題。在蛋白質(zhì)數(shù)據(jù)庫(PDB)中,有四種蛋白無法用MR解決:牛屬甘氨酸N-酰基轉(zhuǎn)移酶(GLYAT)、細(xì)菌氧化還原酶、細(xì)菌表面層蛋白(SLP)和來自真菌平革菌屬金孢子菌屬的分泌蛋白,因此研究人員使用RoseTTAFold重新分析蛋白質(zhì)結(jié)構(gòu)。由于蛋白質(zhì)結(jié)構(gòu)的測(cè)定能夠?yàn)樯锕δ芎蜋C(jī)制提供大量見解,團(tuán)隊(duì)也研究了RoseTTAFold是否也能有這樣的功能。研究人員主要針對(duì)兩組蛋白:目前未知結(jié)構(gòu)的G蛋白偶聯(lián)受體;與疾病相關(guān)的人類蛋白質(zhì)。研究結(jié)果發(fā)現(xiàn),即使沒有已知結(jié)構(gòu)的密切同源物,RoseTTAFold模型在活性和非活性狀態(tài)下也能做到準(zhǔn)確預(yù)測(cè)構(gòu)型。研究人員使用RoseTTAFold工具,從序列信息中預(yù)測(cè)了大腸桿菌蛋白復(fù)合物的結(jié)構(gòu)。圖A中,灰色表示第一條亞基,彩色表示第二條亞基。圖C是RoseTTAFold生成的IL-12R/IL-12復(fù)合結(jié)構(gòu)。論文一作Minkyung Baek希望RoseTTAFold這個(gè)新工具未來能夠造福整個(gè)研究領(lǐng)域。
參考資料:
https://www.nature.com/articles/s41586-021-03819-2
https://science.sciencemag.org/content/early/2021/07/14/science.abj8754