使用 scikit-image 進行圖像處理
點擊上方“小白學視覺”,選擇加"星標"或“置頂”
重磅干貨,第一時間送達
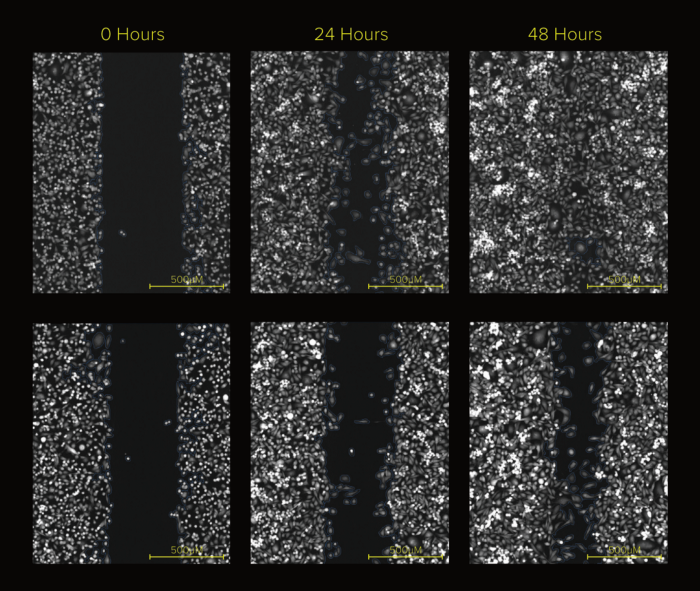
劃痕試驗時間序列分析。
圖像處理在當今非常適用,對于2019冠狀病毒疾病,無論是開發(fā)治療方法還是尋找檢測病毒的新方法,都給全球的研究人員帶來了新的挑戰(zhàn)。為了對 2019冠狀病毒進行研究,研究人員通常需要對圖像進行大量計算,以檢測有助于得出新見解和結論的特定元素。
在這篇文章中,我將介紹圖像處理的基礎知識和一個小教程,詳細介紹如何將圖像處理用于閾值和分割,這是圖像處理中兩種非常強大的技術。在文章中,我們將使用 scikit-image,因此可能需要 Python 的基本背景知識和一些高等數學知識才能完全理解該概念。
除非我們有豐富的生物學經驗,否則大多數人可能不知道什么是劃痕試驗,我將簡要概述什么是劃痕檢測,然后直接進入教程。
劃痕試驗是生物學家用來跟蹤細胞運動和相互作用的一種技術,具體來說,生物學家在培養(yǎng)皿中采集細胞樣本,并在細胞平面上制造人工傷口,在開發(fā)人造傷口后,生物學家會定期拍攝任何細胞運動的圖像,以開發(fā)出一系列用于分析的圖像。
在下面的教程中,我們將處理下面提供的圖像,因此在開始之前下載此圖像并將其添加到我們的項目目錄中。

從顯微鏡獲得的劃痕分析圖像
首先,我們需要確定我們要試圖做什么來處理此圖像,為了從該圖像中獲得更多的信息,我們將使用各種分割技術來分離細胞區(qū)域和劃痕區(qū)域(空白區(qū)域)。如我們所見,由于整個圖像的像素值基本相同,因此我們不能僅根據顏色對該圖像進行分割,但是我們可以使用紋理作為參數來確定培養(yǎng)皿的哪個區(qū)域有細胞,哪個區(qū)域沒有細胞。為此,在開始分割圖像之前,我們將對圖像應用一個特殊的過濾器。
首先,讓我們將所有必要的庫導入我們的 Python 腳本:

然后,導入我們上面提供的圖像,我們在下面的代碼片段中將圖像命名為“scratch.jpg”。

在上面的代碼中,我使用了scikit image中的io包來使用imread函數,該函數允許我輸入圖像數據,此函數的工作原理與用于 CSV 文件的 Pandas read_csv 方法完全相同。此外,我們使用了 scikit-image 的形態(tài)學包中的熵過濾器,這將允許我們通過搜索某些像素中的無序情況來分析圖像的紋理。如果小伙伴們想進一步了解所有這些函數的功能和參數,請訪問 scikit-image 文檔。我們可以使用磁盤參數,但對于此圖像,磁盤值為 10 時可獲得最佳結果。

現在,我們將應用Otsu閾值濾波器,該濾波器來自skiliage.filters包,這個簡單的單行命令將允許我們應用Otsu閾值濾波器。Otsu閾值是一種特定算法,它迭代圖像中的每個像素并將像素分為兩類:背景和前景。一旦我們實際繪制了圖像,我們就會理解其效果。
閾值過濾器只返回單個數字,該值是 Otsu 算法為優(yōu)化圖像分割計算的精確值,將幫助我們生成最終圖像。


最后,在這里我們可以看到圖像分割的結果。在這段代碼中,我們定義了一個子圖,其中將顯示原始圖像、使用熵過濾器的圖像以及同時使用熵過濾器和 Otsu 過濾器的圖像。“帶 Otsu 過濾器的分段試驗”圖像將是我們劃痕試驗分析的最終結果,讓我們看看所有這些代碼的輸出:
正如我們在上面的圖像中看到的,熵過濾器在原始圖像中產生了巨大的變化,熵過濾器檢測圖像像素中的微小變化,并根據圖像某些區(qū)域的復雜性劃分像素。正如我們在第二張圖片中看到的,黑色條帶代表沒有細胞的區(qū)域,而灰色區(qū)域代表有細胞的區(qū)域。Otsu 過濾器通過實現一個閾值來鞏固這種分割,使我們能夠在劃痕分析的準確時刻準確地定位細胞的位置。

最后,我們可以打印代表檢測中無細胞區(qū)域的像素數,以便更好地了解細胞是如何隨著時間移動和增殖的。
這個具體的項目對于理解圖像處理的一些基本方面非常有用,由于所有像素具有相同的 RGB 值,因此在本教程中分析的圖像非常難以分割。然而,在熵過濾器的幫助下,我們能夠生成一個基于紋理處理的圖像,然后我們可以使用 Otsu 閾值來更深入地了解該時間點的細胞位置。
劃痕分析由一個時間序列組成,我們只分析了該時間序列中的一張圖像,生物學家在多個圖像中多次循環(huán)此過程,以生成細胞運動的時間序列表示。當他們?yōu)g覽每張圖像時,生物學家會記錄細胞運動和位置的變化,這為他們提供更多有關細胞增殖率和細胞遷移模式的信息。
許多圖像處理過濾器需要復雜的高斯數學知識,而大多數人沒有這方面的經驗,但是 scikit-image 消除了實現此類過濾器所需的高級數學知識。然而,對高斯核和其他此類計算工具的了解,可以讓研究人員獲得預包裝軟件能夠提供的更準確的可視化效果。
我在下面添加了完整的代碼要點。
In [ ]:from skimage.filters.rank import entropyfrom skimage.morphology import diskfrom skimage.filters import threshold_otsufrom skimage import ioimport matplotlib.pyplot as pltimport numpy as npimport skimageIn [ ]:img = io.imread('images/scratch.jpg') # Reading Imageentropy_img = entropy(img, disk(10)) # Applying entropy filter for better segmentationIn [ ]:thresh = threshold_otsu(entropy_img) # Applying Otsu filter for optimal thresholdingIn [ ]:# Creating image in which all pixels with value less than threshold is equal to truebinary = entropy_img <= thresh # Composed of 0s and 1s (False and True)In [ ]:# Displaying Imagesfig, ax = plt.subplots(nrows=1, ncols=3, figsize=(12,10))ax[0].imshow(img)ax[0].set_title("Original Assay")ax[1].imshow(entropy_img)ax[1].set_title("Assay w/ Entropy Filter")ax[2].imshow(binary)ax[2].set_title("Segmented Assay w Otsu Filter")In [ ]:print(np.sum(binary == 1)) # the number of pixels that correspond to the clear space in the assay
交流群
歡迎加入公眾號讀者群一起和同行交流,目前有SLAM、三維視覺、傳感器、自動駕駛、計算攝影、檢測、分割、識別、醫(yī)學影像、GAN、算法競賽等微信群(以后會逐漸細分),請掃描下面微信號加群,備注:”昵稱+學校/公司+研究方向“,例如:”張三 + 上海交大 + 視覺SLAM“。請按照格式備注,否則不予通過。添加成功后會根據研究方向邀請進入相關微信群。請勿在群內發(fā)送廣告,否則會請出群,謝謝理解~

