在R中使用LIME解釋機(jī)器學(xué)習(xí)模型
作者:PURVAHUILGOL
翻譯:陳丹
校對:歐陽錦
關(guān)鍵詞:機(jī)器學(xué)習(xí)模型解釋、R語言、LIME
概述
僅僅構(gòu)建模型但無法解釋它的輸出結(jié)果是不夠的。
本文中,要明白如何在R中使用LIME來解釋你的模型。
介紹
我曾經(jīng)認(rèn)為花幾個小時(shí)來預(yù)處理數(shù)據(jù)是數(shù)據(jù)科學(xué)中最有價(jià)值的事情。這是我還作為一個初學(xué)者時(shí)的誤解。現(xiàn)在,我意識到,能向一個對機(jī)器學(xué)習(xí)或其他領(lǐng)域的行話不太了解的外行解釋你的預(yù)測和模型才更有意義。
考慮一下這個場景——你的問題陳述涉及預(yù)測患者是否患有癌癥。千辛萬苦,你獲得和清理了數(shù)據(jù)、為它構(gòu)建了模型,并經(jīng)過大量的努力、實(shí)驗(yàn)和超參數(shù)調(diào)整,你達(dá)到了90%以上的精度。太棒了!你走到醫(yī)生跟前告訴他,你可以90%肯定地預(yù)測病人是否得了癌癥。
然而,醫(yī)生問的一個問題會把你難倒:“每個病人都不同,并且會有很多參數(shù)可以決定腫瘤的良惡性,我和病人要怎么相信你的預(yù)測?”
這就是模型可解釋性問題的由來——如今,有多種工具可以幫助你高效地解釋模型和模型預(yù)測,而不必深入探究模型的各種細(xì)節(jié)。這些工具包括SHAP、Eli5、LIME等。今天,我們將討論LIME。
在本文中,我將解釋LIME以及在R中它如何使解釋模型變得容易。
什么是LIME?
LIME全稱是“Local InterpretableModel-Agnostic Explanations”(局部可解釋的模型無關(guān)闡釋)。2016年首次提出LIME技術(shù)的論文被它的作者,MarcoTulio Ribeiro、SameerSingh和CarlosGuestrin,恰當(dāng)?shù)孛麨椤丁癢hyShould I Trust You?” Explaining the Predictions of Any Classifier》(《“為什么我應(yīng)該相信你?”解釋任何分類器的預(yù)測》)。

基于這一基本但至關(guān)重要的信任原則,LIME背后的理念是回答每個預(yù)測和整個模型是“為什么”。LIME的創(chuàng)造者列出了四個必須滿足的基本準(zhǔn)則:
可解釋性:對預(yù)測的解釋應(yīng)該是可以理解的,即可由目標(biāo)人群解釋。
局部準(zhǔn)確性:我們應(yīng)該能夠解釋個體的預(yù)測。作者稱之為“l(fā)ocal fidelity”。
模型無關(guān)性:解釋方法應(yīng)適用于所有模型。這被作者稱為解釋是“model-agnostic”。
整體解釋性:除了個體預(yù)測外,模型還應(yīng)具有整體解釋性,即應(yīng)考慮全局視角。
LIME如何工作?
進(jìn)一步展開LIME的工作原理發(fā)現(xiàn),其背后的主要假設(shè)是,每個模型在局部尺度上都像一個簡單的線性模型,即在單個行級別的數(shù)據(jù)上。即使這篇論文和作者并不打算證明這一點(diǎn),但我們可以感知,在個體水平上,我們可以在行上擬合這個簡單模型,它的預(yù)測將非常接近我們復(fù)雜模型對該行的預(yù)測。很有趣,不是嗎?
此外,LIME還擴(kuò)展了這一現(xiàn)象,即圍繞這一行中的小變化來擬合這些簡單模型,然后通過比較簡單模型和復(fù)雜模型對該行的預(yù)測來提取重要特征。
LIME既適用于表格/結(jié)構(gòu)化數(shù)據(jù),也適用于文本數(shù)據(jù)。
你可以在這里閱讀更多關(guān)于LIME如何使用Python的內(nèi)容
(https://www.analyticsvidhya.com/blog/2017/06/building-trust-in-machine-learning-models/),本文中我們將介紹如何使用R。
所以啟動你的Notebooks或Rstudio,讓我們開始吧!
在R中使用LIME
第一步:安裝LIME和其他所有這個項(xiàng)目所需要的包。如果你已經(jīng)安裝了它們,你可以跳過這步,從第二步開始。
install.packages('lime')install.packages('MASS')install.packages("randomForest")install.packages('caret')install.packages('e1071')
第二步:安裝好這些包后,我們先導(dǎo)入它們:
library(lime)library(MASS)library(randomForest)library(caret)library(e1071)
由于我們用解釋病人是否患有癌癥的預(yù)測作為例子,我們將使用活檢數(shù)據(jù)集。這個數(shù)據(jù)集包含了699名患者及其乳腺癌腫瘤活檢的信息。
第三步:我們導(dǎo)入這些數(shù)據(jù)并看看前幾行數(shù)據(jù):
data(biopsy)
第四步:數(shù)據(jù)勘探
4.1 由于ID列只是一個標(biāo)識符,并沒有用,因此我們首先將它移除:
biopsy$ID<- NULL4.2 讓我們重新命名剩下的列,這樣當(dāng)我們使用LIME來理解預(yù)測結(jié)果的可視化解釋過程中,我們能夠清晰了解特征的名稱:
names(biopsy) <- c('clumpthickness', 'uniformity cell size', 'uniformity cell shape', 'marginaladhesion', 'single epithelial cell size', 'bare nuclei', 'bland chromatin','normal nucleoli', 'mitoses','class')4.3 接下來,檢查是否有缺失值。如果有,在進(jìn)一步處理前,我們應(yīng)先處理它們:
sum(is.na(biopsy))4.4 我們現(xiàn)在有兩種選擇:要么既可以補(bǔ)全這些值,要么也可以使用na.omit函數(shù)直接丟掉包含缺失值的行。由于本文不涉及清理數(shù)據(jù)的內(nèi)容,因此我們將使用后一種方法。
biopsy <-na.omit(biopsy)sum(is.na(biopsy))
最后,讓我們看看前幾行數(shù)據(jù)來確認(rèn)我們的數(shù)據(jù)表。
head(biopsy,5)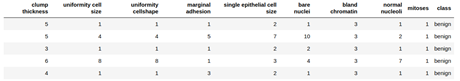
第五步:將這些數(shù)據(jù)分為訓(xùn)練集和測試集,并檢查數(shù)據(jù)的維度。
# 75% of thesample sizesmp_size <-floor(0.75 * nrow(biopsy))## set theseed to make your partition reproducible - similar to random state in Pythonset.seed(123)train_ind<- sample(seq_len(nrow(biopsy)), size = smp_size)train_biopsy<- biopsy[train_ind, ]test_biopsy<- biopsy[-train_ind, ]
檢查維度:
cat(dim(train_biopsy),dim(test_biopsy))因此,在訓(xùn)練集中有512行,測試集中有171行數(shù)據(jù)。
第六步:我們將通過caret包使用隨機(jī)森林模型。我們也不會調(diào)試超參數(shù),只是實(shí)現(xiàn)一個5次10折的交叉驗(yàn)證和一個基礎(chǔ)的隨機(jī)森林模型。所以在我們訓(xùn)練集上訓(xùn)練和擬合模型時(shí),不要進(jìn)行干預(yù)。
我鼓勵你們也可以用這些參數(shù)來試驗(yàn)其他模型。
model_rf <- caret::train(class~ ., data = train_biopsy,method = "rf", #random foresttrControl = trainControl(method ="repeatedcv", number = 10,repeats = 5, verboseIter = FALSE))
讓我們看看模型總結(jié):
model_rf
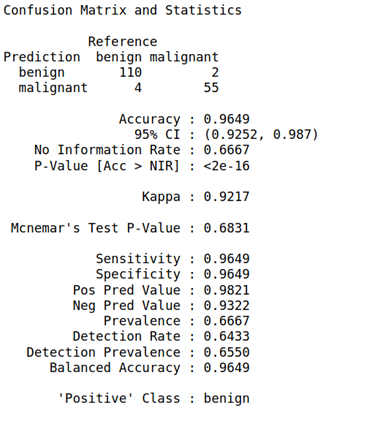
第七步:我們將把預(yù)測函數(shù)運(yùn)用到我們的測試集上,并且建立一個混淆矩陣:
biopsy_rf_pred<- predict(model_rf, test_biopsy)confusionMatrix(biopsy_rf_pred,as.factor(test_biopsy$class))
第八步:有了模型后,我們將用LIME來創(chuàng)造一個“explainer”實(shí)體。這個實(shí)體也與我們將使用來查看解釋的其他LIME函數(shù)有關(guān)。
就像訓(xùn)練模型并擬合數(shù)據(jù)一樣,我們也使用lime() 函數(shù)來訓(xùn)練explainer,然后使用explainer()來得到新的預(yù)測結(jié)果。
explainer<- lime(train_biopsy, model_rf)讓我們只使用5個特征來闡釋從測試集得到的5個觀測值。可以隨意測試任何多個特征參數(shù)。這一步也可以跳過。
整個測試集,或測試集的單一行
explanation <-explain(test_biopsy[15:20, ], explainer, n_labels = 1, n_features = 5)其他你可以試驗(yàn)的參數(shù)有:
1. n_permutation: 用于每次解釋的置換次數(shù)。
2. feature_select: 用于挑選特征的算法。我們可以從以下6個中選擇:
auto:如果n_features小于等于6,則使用“forward_selection”;,否則,使用“highest_weights”。
none:忽略n_features并且使用所有特征。
forward_selection:根據(jù)脊回歸模型的質(zhì)量,每次添加一個特征直到達(dá)到n_features。
highest_weights:擬合一個脊回歸模型并選擇一個n_features作為最高絕對權(quán)重。
lasso_path:擬合一個lasso模型并選擇一個最小角回歸路徑最后收斂到0的n_features。
tree:擬合一個樹來選擇n_features(需要是2的次方)。需要XGBoost的最新版本。
3. dist_fun:距離函數(shù)。我們將使用這個函數(shù)來比較我們的局部模型對某一行的預(yù)測和全局模型(隨機(jī)森林)對該行的預(yù)測。默認(rèn)是高爾距離,但我們也可以使用歐氏距離、曼哈頓距離等等。
4. kernel_width:個體置換預(yù)測和全局預(yù)測的距離如上計(jì)算,并轉(zhuǎn)換成一個相似度評分。
第九步:讓我們來可視化這些解釋以更好地理解它們:
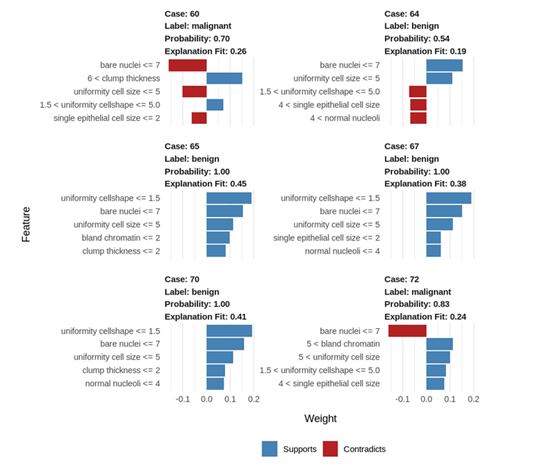
如何解釋這些結(jié)果?
1. 藍(lán)/紅色:正相關(guān)的特征標(biāo)記為藍(lán)色,負(fù)相關(guān)的特征標(biāo)記為紅色。
2. Uniformity cell shape(細(xì)胞形狀的均勻性)<=1.5:更低的值與良性腫瘤正相關(guān)。(值越低,良性腫瘤概率更高)
3. Bare nuclei(裸核)<= 7:更低的值與惡性腫瘤負(fù)相關(guān)。(值越低,惡性腫瘤概率更低)
4. 65、67和70號案例很相似,但是64號良性案例有不一樣的參數(shù)。
5. 在這種情況下,細(xì)胞形狀的均勻性和單個上皮細(xì)胞的大小是異常的。
6. 盡管有這些偏離值,這個腫瘤依舊是良性的,這表明這個病例的其他參數(shù)值補(bǔ)償了這種異常。
讓我們也來可視化一個案例的所有特征:
explanation <-explain(test_biopsy[93, ], explainer, n_labels = 1, n_features = 10)plot_features(explanation)
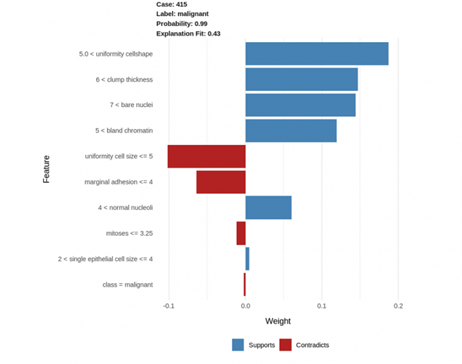
Uniformity cellshape(細(xì)胞形狀的均勻性) > 5.0:高的值與惡性腫瘤正相關(guān)(這個值越高,腫瘤惡性的可能性更大)。
Clump thickness(團(tuán)塊厚度) > 6.0:高團(tuán)塊厚度值與惡性腫瘤正相關(guān)。
同樣的,bare nuclei(裸核) > 7.0 以及 bland chromatin(布蘭德染色質(zhì))> 5.0 與惡性腫瘤正相關(guān)。
相反,uniformity of cell size(細(xì)胞大小的均一性)<= 5.0 和marginaladhesion(邊緣附著力)<= 4:這2個參數(shù)低值與惡性腫瘤的惡性成正相關(guān)與惡性腫瘤負(fù)相關(guān)[1] 。因此,這些值越低,腫瘤惡性的可能性越小。
因此,從上可知,我們可以得出結(jié)論,這些參數(shù)高值能夠表明腫瘤有更高可能性是惡性。
我們也可以通過看這行真實(shí)的數(shù)據(jù)來確定上述的解釋:

尾注
最后,我們探討了LIME以及如何使用它來解釋我們模型的個體結(jié)果。這些解釋更好地幫助我們傳達(dá)自己的想法有助于更好地講故事,并幫助我們向一個可能擁有領(lǐng)域?qū)I(yè)知識,但沒有模型構(gòu)建技術(shù)訣竅的人解釋為什么模型會做出某些預(yù)測。而且,使用它非常容易,在我們有了最終的模型之后只需要幾行代碼。
然而,這并不是說LIME沒有缺點(diǎn)。我們使用的LIME-Cran包并不是我們在本文中介紹的原始Python實(shí)現(xiàn)的直接復(fù)刻,因此,它不像Python那樣支持圖像數(shù)據(jù)。另一個缺點(diǎn)是,局部模型可能并不總是準(zhǔn)確的。
我期待著使用不同數(shù)據(jù)集和模型來更多地探索LIME,并且探索R中的其他技術(shù)。你在R中使用了哪些工具來解釋你的模型?一定要在下面分享你如何使用他們以及你使用LIME的經(jīng)歷!
這里應(yīng)該也是正相關(guān)吧。值越低,越不容易產(chǎn)生惡性腫瘤。
原文標(biāo)題:
PolynomialRegression — Gradient Descent from ScratchMLInterpretability using LIME in R
原文鏈接:
https://www.analyticsvidhya.com/blog/2021/01/ml-interpretability-using-lime-in-r/
推薦閱讀:
用戶畫像、用戶標(biāo)簽和用戶分群有什么區(qū)別和聯(lián)系呢?
用python分析《三國演義》中的社交網(wǎng)絡(luò)
給新手:學(xué)數(shù)據(jù)分析沒數(shù)據(jù)練習(xí),推薦一款工具給你
更多精彩內(nèi)容,請?jiān)L問【大數(shù)據(jù)科學(xué)】小程序
譯者簡介
陳丹,復(fù)旦大學(xué)大三在讀,主修預(yù)防醫(yī)學(xué),輔修數(shù)據(jù)科學(xué)。對數(shù)據(jù)分析充滿興趣,但初入這一領(lǐng)域,還有很多很多需要努力進(jìn)步的空間。希望今后能在翻譯組進(jìn)行相關(guān)工作的過程中拓展文獻(xiàn)閱讀量,學(xué)習(xí)到更多的前沿知識,同時(shí)認(rèn)識更多有共同志趣的小伙伴!

